для чего нужен секвенатор
Нанопоровое секвенирование: на пороге третьей геномной революции
Нанопоровое секвенирование является одной из наиболее перспективных современных технологий определения последовательности нуклеиновых кислот, позволяя решать многочисленные задачи, стоящие перед биологами
Автор
Редакторы
Публикация первого генома человека в 2001 году стала предвестником постгеномной эры — появление технологий секвенирования нового поколения (next-generation sequencing, NGS) позволило поверить в будущее персонифицированной геномики. Сегодня, спустя более 15 лет, коммерциализация приборов, чья работа основана на нанопоровом секвенировании, делает это будущее реальностью. Давайте же обсудим, чем так привлекательна новая технология.
Партнер публикации этой статьи — компания «Диаэм»: крупнейший поставщик оборудования, реагентов и расходных материалов для биологических исследований и производств.
Секвенирование ДНК — рутинная процедура, позволяющая определять нуклеотидную последовательность этой макромолекулы. Современные методы полногеномного секвенирования делают возможным считывать миллиарды последовательностей ДНК за считанные часы — буква за буквой, ген за геном и хромосома за хромосомой. Уже отсеквенированы десятки тысяч геномов, начиная от вирусных и заканчивая геномами человека, полиплоидных и даже вымерших видов. Всё это происходит благодаря новейшим полногеномным технологиям, о которых я написал и опубликовал на «Биомолекуле» большой обзор: «12 методов в картинках: секвенирование нуклеиновых кислот» [1]. Поэтому, если вы желаете поглубже окунуться в дебри современного, и не очень, секвенирования, рекомендую обратиться сначала к нему.
Но время, впрочем как и развитие технологий, не стоит на месте, и в жизни каждого из нас нередко встает проблема, для решения которой необходимо перелопатить десятки публикаций и книг. Совсем недавно с подобными сложностями столкнулись я и мои коллеги из лаборатории палео- и этногенетики НИЦ «Курчатовский институт» в попытке впервые в мире собрать полиплоидный геном одного, находящегося на грани вымирания, организма, который ни в какую не хотел объединяться в единое целое. Знающие люди посоветовали нам присмотреться к нанопоровому секвенированию — решению, позволяющему прочитывать длинные молекулы ДНК за приемлемые деньги. Недолго думая, мы решили разобраться, какие же преимущества сулит эта относительно новая технология, и пробежались по уже опубликованным работам, оценив весь спектр предложений геномных секвенаторов Oxford Nanopore Technologies, поставляемых на российский рынок компанией «Диаэм».
Нанопоровое секвенирование — история и современное состояние. Плюсы и минусы
Знакомясь с историей появления нанопорового секвенирования, можно еще раз убедиться в необходимости как разумного государственного финансирования фундаментальной науки, так и венчурных инвестиций в идеи и прототипы, неожиданно прорезающиеся посреди суровых формул, запутанных биохимических реакций и сложных молекулярных структур.
Первые шаги к созданию технологии нанопорового секвенирования были сделаны более двадцати лет назад, когда группа американских ученых, используя напряжение электрического поля, научилась «протаскивать» молекулы ДНК и РНК — как нитку через игольное ушко — сквозь ионный канал диаметром 2,6 нанометра, закрепленный в бислойной липидной мембране. Наверное, на переломе XX и XXI веков эта работа могла бы показаться обычным физико-химическим методом изучения макромолекул, но уже тогда исследователи сумели различать молекулы ДНК и РНК, а также оценивать их длину [2]. В 2001 году, используя этот подход, стало возможным отличать друг от друга короткие одноцепочечные ДНК, содержащие отдельные нуклеотидные замены [3], а уже в 2009 году нанопоровое секвенирование стало явью — сотрудники Оксфордского университета научились различать все четыре нуклеотида в одноцепочечной ДНК по мере их прохождения через пору [4], что предвосхитило появление первого секвенатора от компании Oxford Nanopore Technologies в 2014 году.
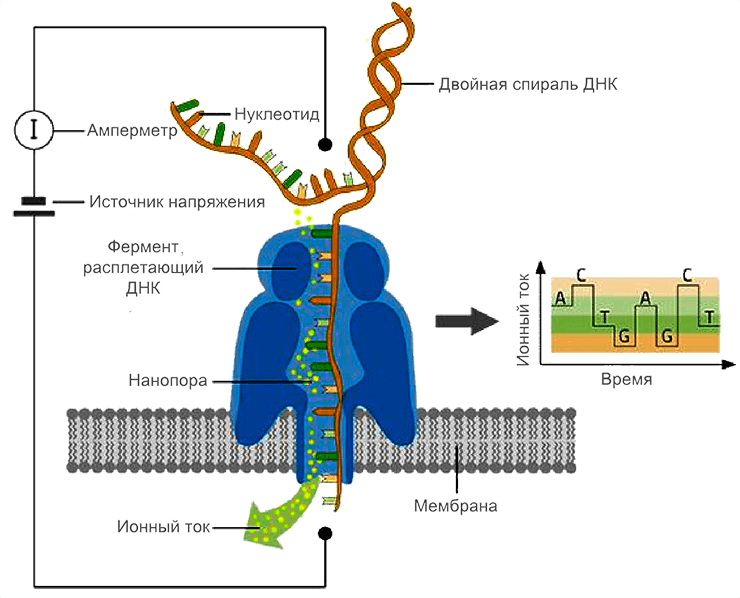
Технология, предложенная британской компанией и внедренная в её секвенирующие устройства, достаточно проста. Молекула ДНК или РНК, несущая на себе существенный отрицательный заряд, под воздействием электрического поля «протискивается» через белковую пору, помещенную в бислойную мембрану, что вызывает уменьшение силы электрического тока из-за уменьшения сечения отверстия (рис. 1 и видео). Считывая изменение силы тока, нанопоровый секвенатор определяет нуклеотид, проходящий через пору в конкретный отрезок времени: оказывается, что каждый «кирпичик» в нуклеиновой кислоте имеет свое собственное «сечение» (и, соответственно, это влияет на силу тока).
Рисунок 1. Белковая нанопора, закрепленная в бислойной мембране, позволяет считывать десятки тысяч «букв» за один запуск секвенатора
Видео. Принцип работы нанопорового секвенатора
Методика, реализованная в устройствах MinION, GridION X5, PromethION и SmidgION, как и любая другая, имеет и плюсы, и минусы. В первую очередь, от нее не следует ждать такой же производительности, как от последних версий геномных «фабрик», выведенных на биотехнологический рынок компанией Illumina: ведь степень параллелизма в нанопоровом секвенировании гораздо ниже, да и само «протискивание» через нанопору — процесс пока не настолько производительный. Однако современные приборы от Oxford Nanopore имеют и сильные стороны.
В первую очередь это длина прочтений, которая может достигать 100 тысяч нуклеотидов и представляется недостижимой для других технологических платформ. Так, многие коммерциализированные к настоящему времени методики секвенирования «нового поколения» основываются на многократном и одновременном прочтении коротких последовательностей ДНК (до 300 нуклеотидов), что позволяет с одной стороны значительно ускорить сложный ферментативный процесс, а с другой — значительно увеличить копийность (глубину покрытия генома). Данные, которые исследователи получают в результате такого секвенирования, больше походят на «геномный паззл», а не на разложенный по хромосомам геном.
Длина прочтения в десятки тысяч нуклеотидов, предлагаемая нанопоровым секвенированием, позволяет не только значительно упростить и ускорить сборку ранее не опубликованных геномов, но и улучшить уже существующие сборки. Следует особо отметить, что такие секвенаторы не испытывают «аллергии» на участки с повышенным содержанием гомополимеров и на GC-богатые области генома, в отличие от многих альтернативных технологий.
Не секрет, что не все геномы собраны настолько же идеально, как, допустим, человеческий или мышиный. Большинство из них, по сути, представляют собой великое множество длинных фрагментов (контигов), расположение которых на хромосоме неизвестно, поэтому доступность длинных прочтений значительно упростит процесс de novo сборки генома, который состоит из сложения единичных фрагментов в цельную последовательность [5].
Кроме того, нанопоровое секвенирование при достаточном количестве предварительно экстрагированного из биологического образца генетического материала позволяет отказаться от ПЦР : отсутствие стадии амплификации ДНК при пробоподготовке помогает избежать выборочного обогащения определенных участков генома, а также устраняет необходимость длительной и не самой простой пробоподготовки, например, при изучении модификаций нуклеотидов в геноме [8]. Отсутствие стадии ПЦР и упрощение используемых реагентов делают исследователя более мобильным и значительно удешевляет процесс пробоподготовки и секвенирования. Размер нанопоровых секвенаторов делает их крайне утилитарными устройствами, позволяя проводить исследования в самых неожиданных местах — Экваториальной Африке, Антарктиде или в космосе, — буквально, «на коленке»: маркетологи не преминули воспользоваться этим преимуществом, именуя самый миниатюрный из них (MinION) секвенатором-флешкой (рис. 2).
Что кроется за аббревиатурой «ПЦР», вы можете узнать из нашего обзора «Полимеразная цепная реакция», входящего в цикл «12 биологических методов в картинках» [9]. — Ред.
Рисунок 2. Нанесение пробы на проточную ячейку секвенатора MinION
Неоспоримым преимуществом нанопорового секвенирования является также анализ полученных данных в режиме реального времени — исследователь в любой момент запуска может понять, что процесс идет не совсем так, как хотелось бы, остановить секвенирование и нанести на проточную ячейку секвенатора новый образец.
Оценивая плюсы и минусы платформы, можно смело сказать, что у нанопорового секвенирования неплохое будущее на мировом биотехнологическом рынке, где оно может уже сегодня найти применение и как дополнительная, и как самостоятельная секвенирующая платформа. Несмотря на очевидно высокий процент ошибок, небольшая цена приборов и реагентов к ним, простота использования, а также уникально длинные прочтения сделают этот девайс безусловным лидером продаж, а также подтолкнут компанию Oxford Nanopore к дальнейшему развитию линейки.
Современные приборы нанопорового секвенирования Oxford Nanopore Technologies
Технология нанопорового секвенирования, реализованная в приборах, поставляемых британской компанией Oxford Nanopore Technologies (официальный дистрибьютор в России и СНГ — компания «Диаэм»), по своему принципу кардинально отличается от уже существующих платформ, а также славится своей универсальностью, компактностью и простотой использования.
Так, простейший секвенатор MinION весит каких-то сто граммов и работает от USB 3.0 настольного компьютера, позволяя, например, секвенировать за один запуск геном человека с шестикратным покрытием.
Для большей производительности Oxford Nanopore Technologies разработал приборы, в которые можно параллельно установить сразу несколько проточных ячеек (продуктовая линейка изображена на рисунке 3):
Рисунок 3. Линейка геномных секвенаторов Oxford Nanopore Technologies, поставляемых на российский рынок компанией «Диаэм»
Кроме самих секвенаторов при работе со значительным количеством образцов компания Oxford Nanopore Technologies рекомендует дополнительно приобрести автоматизированную станцию для пробоподготовки VolTRAX, позволяющую полностью отстраниться от этого процесса. VolTRAX берет на себя все заботы по приготовлению биологических образцов к секвенированию, начиная от выделения ДНК или РНК из ткани и заканчивая приготовлением ДНК-библиотек.
Опытные геномщики, конечно, зададутся вопросом обсчета данных, но и тут ожидает приятный сюрприз — с 2018 года им не придется подбирать ноутбук с необходимыми параметрами или по крупицам собирать мощный настольный компьютер; для удобства своих пользователей Oxford Nanopore Technologies анонсировал продажу специального миниатюрного (менее 300 граммов), но крайне производительного портативного компьютера MinIT, позволяющего обрабатывать данные в любой точке мира.
Применение нанопорового секвенирования
Продолжая свое знакомство с нанопоровым секвенированием и приборами, использующими эту технологию, я не преминул оценить возможные перспективы внедрения приборов компании Oxford Nanopore Technologies в научную работу, одним из направлений которой является сборка геномов видов животных, находящихся на грани уничтожения. Для этого мы пробежались по статьям, использующим эту технологическую платформу.
В погоне за патогеном
В первую очередь новой платформой заинтересовались исследователи, занимающиеся геномикой микроорганизмов. Лишенные длинных прочтений секвенаторами GS FLX и 454 [10] производства Roche (поддержка платформы остановилась в 2016 году), они выбирают длинные фрагменты, генерируемые нанопоровыми секвенаторами, которые при многократном прочтении и использовании специального биоинформатического инструментария позволяют снизить процент ошибок (хотя и с некоторым допущениями) до оптимального, в том числе при комбинации этой технологической платформы с Illumina [11], [12].
В то же время понятно, что приборы Oxford Nanopore Technologies имеют огромный потенциал и уже сейчас могут применяться для быстрой идентификации вирусных и бактериальных патогенов [13], [14], а также для анализа в реальном времени сложных метагеномных образцов из окружающей среды, причем в самых необычных условиях — на антарктической станции и даже в космосе [15], [16].
Нанопора и альтернативный сплайсинг
Процесс «дозревания» молекул РНК, в результате которого из пре-мРНК (предшественника мРНК) образуется несколько зрелых мРНК. — Прим. авт.
Преимущества для de novo сборки
Нельзя также не отметить возможность использования длинных прочтений при работе с de novo собранными геномами или транскриптомами, и пусть до похромосомной сборки еще далековато, но комбинация длинных прочтений с данными традиционных NGS-платформ позволяет увеличивать параметры сборки на порядки [18–20]. Нанопоровое секвенирование проникает и в Россию: так, исследователи биологического факультета Санкт-Петербургского государственного университета уже заявили о значительном улучшении качества и размера сборки генома пресноводной мшанки Cristatella mucedo при комбинировании данных стандартного NGS и нанопорового секвенирования.
Медицина и криминалистика
Быстрое и относительно продуктивное секвенирование в реальном времени сулит замечательные прорывы не только в фундаментальных исследованиях, но и в медицине и биотехнологии, поскольку позволяют оперативно решать насущные задачи — например, проводить предимплантационную генетическую диагностику эмбрионов [21] или судебно-криминалистическую экспертизу [22].
Загадки эпигенома
Кроме собственно определения последовательности нуклеиновых кислот, нанопоровое секвенирование способно оценивать степень модификации нуклеотидов в геноме, выявляя метилирование ДНК — присоединение метильной группы (—CH3) к цитозину в составе CpG-динуклеотида. В частности, приборы от Oxford Nanopore Technologies позволяют идентифицировать метилированные азотистые основания 5-метилцитозин и 5-гидроксиметилцитозин, таким образом значительно облегчая эпигеномные исследования [23].
Один из способов регуляции работы генома без изменения нуклеотидной последовательности ДНК. Больше об эпигенетике вы узнаете из статей «Эпигеном: параллельная реальность внутри клетки» и «Эпигенетика: невидимый командир генома» на «Биомолекуле» [24], [25]. — Ред.
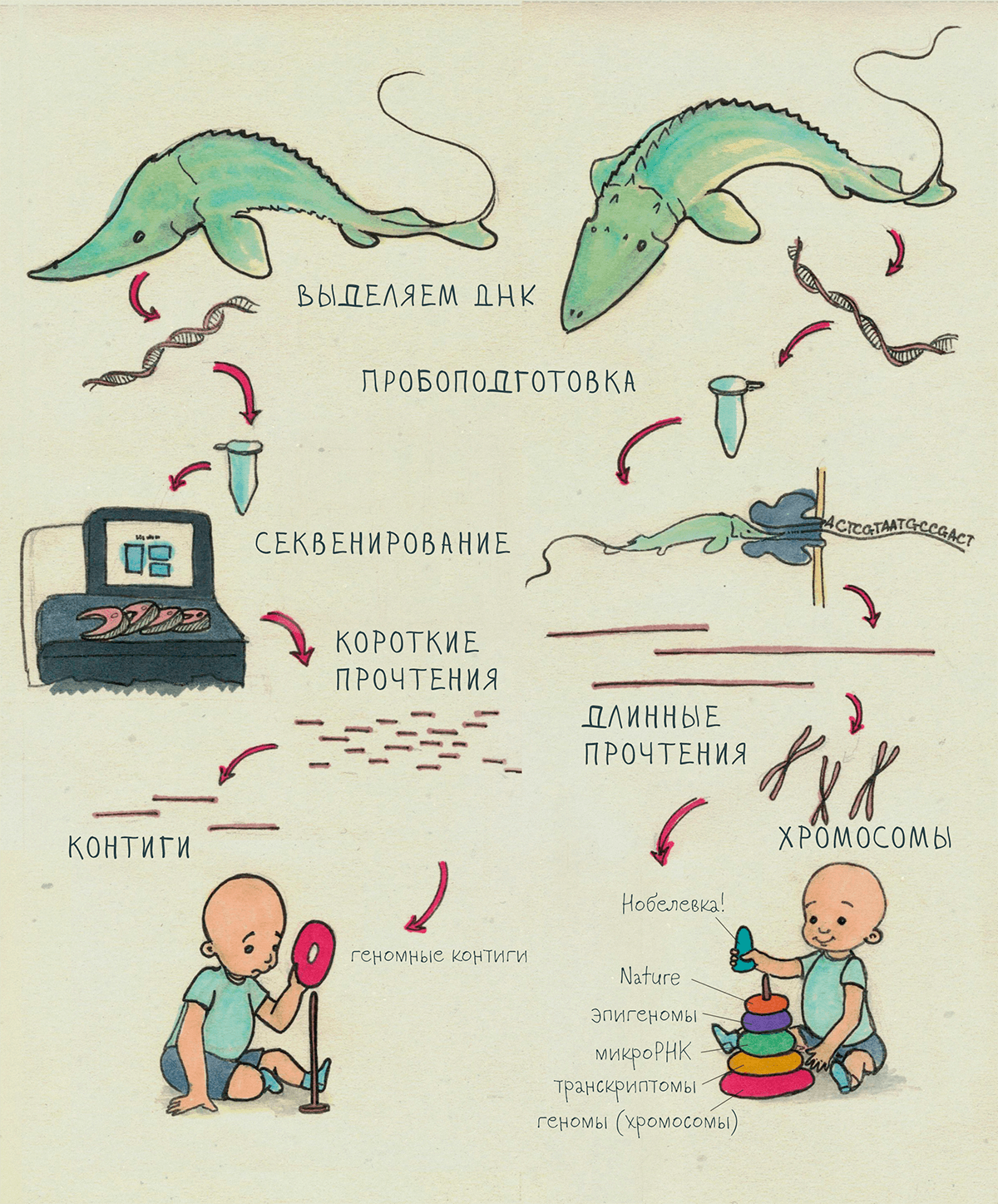
Рисунок 4. Метафорическое сравнение «обычного» NGS-секвенирования (слева) и нанопорового секвенирования «третьего поколения» (справа). Главное преимущество «нанопоры» — длина прочтений, дающая исследователю что-то более-менее похожее на «готовый» геном, нежели «мозаику» контигов. Это сулит возможность «сборки» геномов тех организмов, до которых раньше просто не удавалось добраться (например, полиплоидные осетровые, занесенные в списки охраняемых видов, СИТЕС).
Секвенирование теперь доступно каждому!
Технология секвенирования Oxford Nanopore Technologies позволяет делать прямое прочтение цепей ДНК или РНК в режиме онлайн, длина рида ограничена только длиной фрагмента, а портативность оборудования и быстрая подготовка библиотек дает возможность секвенировать даже в полевых условиях с минимальными требованиями к генетической лаборатории. С Oxford Nanopore Technologies секвенировать теперь может каждый, даже тот, кто ранее и не задумывался о секвенировании — это просто и доступно.
Секвенирование третьего поколения не заменяет и не отменяет применение капиллярных секвенаторов по Сэнгеру или платформ NGS второго поколения. Наоборот, сочетание трех поколений генетического анализа открывает новые возможности получения ранее неизвестных данных.
Специалисты «Диаэм» прошли обучение в Oxford Nanopore Technologies, осуществляют профессиональное консультирование и техническую поддержку, помогают спланировать эксперимент и подобрать необходимые наборы реагентов для решения конкретной задачи независимо от бюджета лаборатории.
Материал предоставлен партнёром — компанией «Диаэм»
Будущее нанопорового секвенирования
Сегодня нанопоровое секвенирование — одна из наиболее перспективных современных технологий определения последовательности нуклеиновых кислот и имеет ряд преимуществ перед традиционными секвенирующими платформами, прочно укоренившимися на биотехнологическом рынке. Упрощенная оценка эпигенетических особенностей генома, длинные прочтения и оценка сложно читаемых участков генома позволяют с оптимизмом смотреть на её дальнейшее развитие.
Имеющиеся недостатки (невысокое качество прочтения генетической информации и относительно невысокий объем данных, получаемых за один запуск прибора), по-видимому, в ближайшие годы будут решены, и тогда будущее действительно станет реальностью.
Взвесив все «за» и «против», оценив все плюсы и минусы нанопорового секвенирования, наша команда также решила влиться в число активных пользователей новой технологии, о результатах использования которой вы узнаете первыми со страниц научных журналов и, конечно же, любимой всеми нами «Биомолекулы».
Секвенирование ДНК в домашних условиях: как на коленке собрать прибор за 10 миллионов
Всем привет, меня зовут Александр Соколов, и я хочу рассказать, как сделал дома секвенатор – прибор для расшифровки ДНК. Рыночная цена такого прибора составляет около 10 миллионов рублей.
Краткий экскурс в генетику. Если вдруг вы помните, в 2003 г. было сделано сенсационное заявление: ученые, наконец, расшифровали геном человека. Геном построен из ДНК, а ДНК – это исходный код организма. ДНК представляет собой двойную цепочку, состоящую из 4-х видов нуклеотидов, которые повторяются в геноме человека порядка 3 млрд. раз. Так же, как в битах зашифрована вся информация на вашем компьютере, в нуклеотидах зашифрована инструкция о сборке всех белков человеческого тела. То есть зная, в какой последовательности расположены нуклеотиды в ДНК, мы теоретически можем собрать все необходимые белки и получить модель человека. Так вот в стандартном понимании ученые не расшифровали ДНК, а просто перевели химическую последовательность в набор нулей и единиц на компьютере. Что делать с этим дальше – отдельный разговор. Например, на данный момент нам ясна функция лишь 5% всего массива генома (это кодирование белков). Чем занимаются остальные 95%, можно только предполагать.
В 2003 году стоимость секвенирования ДНК человека составляла около 100 млн долларов. С течением времени эта цифра уменьшалась и сейчас она приближается к тысяче долларов. Вы платите, вашу ДНК секвенируют и отдают вам жесткий диск с 3 ГБ информации – вашим геномом в цифровом виде.
Что ж, десяти миллионов у меня не было, а PGM захотелось. Пришлось сделать самому. Сначала вкратце о том, как происходит полупроводниковое секвенирование. Вся цепочка ДНК делится на фрагменты длиной по 300-400 нуклеотидов, называемые ридами. Затем риды прикрепляются к маленьким сферам и многократно копируются – в итоге на каждой сфере «висит» целый пучок одинаковых фрагментов ДНК. Копирование нужно для усиления сигнала от каждого конкретного рида. Набор разных сфер называется библиотекой ДНК.
Сердцем PGM является одноразовый чип – матрица, похожая на матрицу в фотокамере, только вместо пикселей, реагирующих на свет, здесь pH-транзисторы, реагирующие на изменение кислотно-щелочного баланса. Полученная библиотека ДНК загружается на чип, содержащий 10 млн лунок, на дне каждой из них находится pH-транзистор. В лунку умещается только одна сфера и, следовательно, риды только одного типа (с одной определенной последовательностью нуклеотидов). Далее на чип подаются реагенты таким образом, чтобы ДНК начала себя копировать. А копируется она линейно, то есть нуклеотиды прикрепляются к вновь создаваемой цепочке в том порядке, в котором они стоят в материнской цепочке. Поэтому на чип подается один тип нуклеотидов – и тут же фиксируется изменение pH в некоторых лунках (это значит, что в них произошло присоединение данного нуклеотида). Далее подается другой тип нуклеотидов и фиксируется изменение pH в лунках и т. д. Таким образом, подавая на чип все 4 типа нуклеотидов много раз, мы можем получить информацию о последовательности нуклеотидов в каждом риде. Затем математическими способами прочитанные короткие отрезки собираются на компьютере в единую цепочку. Чтобы собрать ее более-менее уверенно, каждый рид нужно прочесть примерно по 100 раз.
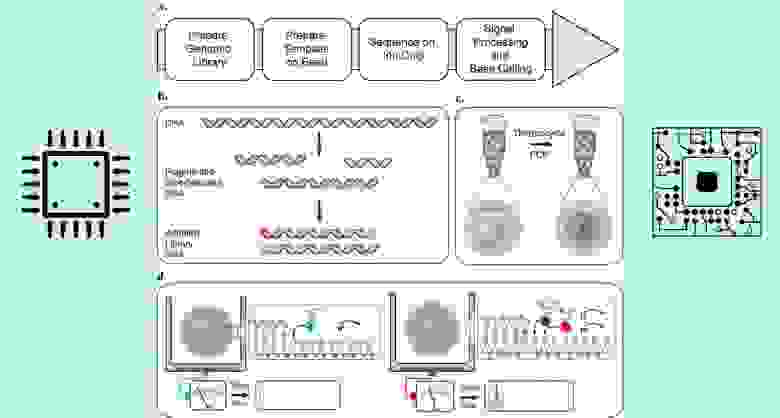
Рис.1. Полупроводниковое секвенирование
Теперь разберемся, из чего состоит сам прибор. Имеется, как мы уже знаем, чип, а также система подачи реагентов и материнская плата. Все секвенирование ведется именно на чипе – остальной аппарат только передает на него определенные сигналы, подает реагенты, считывает с него аналоговые сигналы, оцифровывает их и гонит полученный поток информации на компьютер, где данные накапливаются и обрабатываются.

Рис. 2. Устройство секвенатора
Чип позиционируется как одноразовый и после использования выкидывается. Соответственно, там, где работает PGM, такие чипы можно достать бесплатно в любом количестве. Зачем их доставать, спросите вы? Дело в том, что чип мне уже удалось использовать многократно. По сути он вечен: достаточно хорошо промывать его – и можно применять вновь и вновь. По точности работы он ничем не будет отличаться от нового. Сама моя идея заключалась в том, чтобы сделать прибор под этот условно бесплатный чип.

Рис. 3. Прозвонка чипа
А затем внезапно на Habrahabr мне попалась статья известного блоггера BarsMonster о том, как он делает реверс-инжиниринг чипов! Воодушевился, написал ему, написал другим энтузиастам, отправил запрос в Киев, где занимались фотографированием чипов. Из Киева ответили, что полировать по слоям они не умеют, могут только отснять верхний слой, а так как мой чип – многослойный, будет не понятно, куда идут дорожки от контактов. Потом познакомился с одним американцем, который тоже занимается реверс-инжинирингом чипов, послал ему свои микросхемы, но и тут дальше фотографирования верхнего слоя дело не пошло. Затем наткнулся в интернете на статью про тех, кто смог отреверсить чип Sony PlayStation и пр. («Слава героям!» и вот это все, если кто в курсе). Решил написать им с вопросами, нашел их ники – и тут же понял, что один из них мне знаком. Недавно товарищ свел меня со своим другом, который «тоже занимается генетикой на любительском уровне», мы пообщались с этим другом в Skype и на этом диалог закончили. И вот я понимаю, что мой новый приятель – мегакрутой мастер реверс-инжиниринга чипов. Тут же написал ему. Однако выяснилось, что, хоть помочь он и готов, у него нет микроскопа. Снова тупик.
А через несколько месяцев нужный микроскоп нашелся в соседней лаборатории! Правда, встроенная в него камера была ужасной, я фотографировал на мобильный телефон через окуляр и получал снимки вот такого качества:

Рис. 4. Чип под микроскопом
Затем на последний Новый год отличный микроскоп за 130 тыс. появился у меня на работе (я – специалист по квантовой криптографии). Мечты сбываются. Наконец, я смог нормально сфотографировать чип сверху.
Рис. 5. Мой рабочий микроскоп
А потом… Потом мне все-таки пришлось самому освоить технику его полировки. Трудность полировки заключается в том, чтобы снимать слои металла толщиной порядка 1 микрона – при этом ширина чипа составляет 1 сантиметр. Для сравнения скажу, что это примерно то же, что допустить на 1 км погрешность не более 10 см. Я очень старался. Результаты моих трудов представлены на следующем фото:

Рис. 6. Реверс-инжиниринг под оптическим микроскопом
Довольно хорошо видны нижний кремниевый слой, верхний слой с транзисторами, первый, второй, третий и четвертый слои металла.
Чип состоит из повторяющихся зон (типа сдвиговых регистров), и по таким картинкам было очень удобно его анализировать: сразу становилось ясно, что происходит на разных слоях. Я «отреверсил» самые «нафаршированные» участки с обилием логики, которые многократно повторялись. Но самым сложным оказалось отследить трассы, идущие по всему чипу, понять, какой внешний контакт к чему относится. С новогодних праздников до конца февраля, я, вооружившись новым прекрасным микроскопом, корпел над этой задачей – сидел на работе до десяти ночи, «реверсил», думал. И тут произошло новое чудо: товарищ смог организовать бесплатную фотосъемку чипа по слоям на электронном микроскопе в МИРЭА. «Фотосессия» крохи в 1 кв. см представляла собой 50 ГБ черно-белых фотографий.
Теперь все эти отдельные фотографии нужно было каким-то образом объединить в одну целую картинку. Чуть ли не в тот же день я написал на «питоне» программу, которая генерировала HTML-файл – при его открытии в браузере я получал требуемое. (Кстати, самая старая 10-я Opera справилась с этим лучше всего, рекомендую!) Затем на javascript написал еще одну программу, позволяющую сравнивать слои, плавно переходить между ними, выравнивать их, подбирать масштаб и т. д. Наконец, в моих руках были все инструменты для решения главных задач. Я отследил трассы, пронизывающие чип, и восстановил всю его структуру до последнего транзистора.
Еще одна фотография среза чипа, сделанная под рентгеном (в МИРЭА):

Рис. 7. Съемка под электронным микроскопом
Хорошо видны лунки, куда попадают сферы с ридами. Ниже располагаются три слоя металла, а еще ниже – слой с транзисторами.
Следующим этапом борьбы за светлое будущее стало создание под чип материнской платы. Спроектировал ее и отправил заказ на производство. А пока суд да дело использовал для работы с чипом плату «Марсоход-2» с FPGA. (FPGA – это, грубо говоря, массив из 10 000 универсальных логических элементов; программируя FPGA, мы можем получать любую логическую схему, легко обрабатывающую гигабитные потоки информации.) Прошивку для FPGA я написал сам, а кроме того, для динамического управления системой написал софт, который задает всю конфигурацию для FPGA. Потом вновь образовался полугодовой перерыв (ездил в командировку на Байкал, готовил в лаборатории установку, которую демонстрировали Путину). Но в конце концов звезды сошлись: у меня появилось время, приехали готовые платы – и я собрал свою систему.

Рис. 8. Создание «железа»
Подал все необходимые сигналы и – о, чудо! – увидел на осциллографе сигнал с чипа. (Осциллограф я купил когда-то за 6 000 рублей на eBay, еще 1 000 стоила прошивка к нему.) На картинке хорошо видны пятна – капельки какого-то реагента.
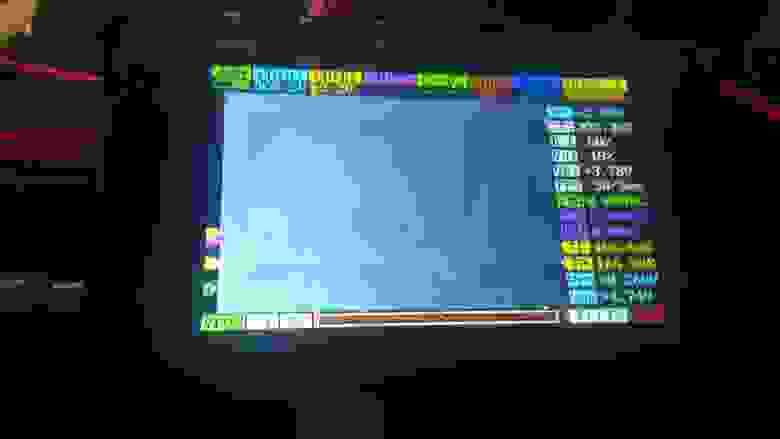
Рис. 9. Сигнал с чипа на осциллографе
Теперь мне нужно было придумать, как оцифровать эту картинку и передать ее на компьютер. Я собрал вот такую установку:
Рис.10. Схема прибора

Рис. 11. Готовая установка
Есть компьютер, который подает данные управления на плату с FPGA. Плата генерирует цифровые сигналы и отправляет их на чип. Сигнал с чипа идет на усилитель, далее – на АЦП на плате, оцифровывается и передается через COM-порт на компьютер. Вообще, пропускная способность COM-порта невелика: 15 килобит в секунду (т. к. в одном чипе находится от 1 млн до 10 млн «пикселей», а максимальная скорость передачи – 115200 бод). Тем не менее картинка на компьютер в итоге попадает.

Рис. 12. Обработанный сигнал на компьютере.
На фото выше видно, что, когда на использованный б/у-шный чип подается библиотека ДНК, чип заполняется неравномерно: по краям – в меньшей степени. Разные цвета обусловлены разным напряжением на pH-транзисторах. То есть мы можем ясно различить те лунки, куда попали сферы с ридами – впоследствии это поможет нам контролировать промывку чипа.
Соответственно, следующей задачей стала промывка чипа. Нужно было добиться, чтобы он стал, как новый. К счастью, у меня имелся совершенно новый чип в качестве референсного образца. На илл. А видно, что в активной области такой чип практически одного цвета (вертикальные повторяющиеся полосы – это просто шумы, наводки).

Рис. 13. Промывка чипа
На рис. 13 B неудачно промытый чип – он разноцветный. На рис.13 D – использованный, но хорошо промытый чип. Видно, что градиент по краям исчез. Тем не менее стоило бы еще доказать, что он действительно чистый и может использоваться повторно.
Поскольку библиотеки ДНК прикрепляются к танталовому покрытию чипа в кислой среде и открепляются – в щелочной (то есть при высоком pH), то чип промывается с помощью специальных полуавтоматических пипеток растворами с разными pH. На сегодняшний день мне удалось добиться практически полной очистки чипа.
У меня интересовались, почему, когда я полностью разобрался в структуре чипа, я не стал заказывать его изготовление, а предпочел по-прежнему искать и доставать б/у-шные, возиться с их промывкой и т. п. Да потому, что разработка микросхемы стоит огромных денег, миллионы долларов, и солидная часть этой суммы уходит на физическую отладку полученного продукта: подгонку, настройку всех параметров транзисторов и т. д. То есть просто скопировать логическую схему – недостаточно. Поэтому я беру условно бесплатную, уже готовую – спроектированную, изготовленную, отлаженную – микросхему и таким образом экономлю значительные средства, серьезно удешевляю проект.
Следующей моей задачей было собрать более продвинутый прибор, который позволял бы быстрее передавать информацию на компьютер и при этом не состоял бы из огромного количества отдельных плат.

Рис.14 Разработка следующей версии прибора
Я взял новую плату с FPGA – на том же кристалле здесь было 2 ARM-ядра с Linux, имелся Gigabit Ethernet и прочие «плюшки», но зато, в отличие от предыдущего варианта, не было АЦП. Позже спроектировал еще одну плату, с высокоскоростными АЦП и всеми другими необходимыми элементами. Запустил – все заработало.
Что осталось сделать для появления финального прибора? Всего три вещи.
Первое. Нужен гигабитный интернет, быстрая передача данных на компьютер. Это я реализовал буквально вчера.
Второе. Система подачи реагентов. Проектирование специального клапана уже в процессе.
Третье. Софт для обработки информации с чипа. С ПО пока есть вопросы, поэтому приглашаю к сотрудничеству программистов.
Финальный прибор стоит 10 млн рублей. Себестоимость секвенирования составляет несколько тысяч долларов. Чипы обходятся от 100 до 1000 долларов – в зависимости от количества «пикселей» в них. (К слову, восстановление чипов само по себе может стать неплохим заработком, особенно учитывая, что для промывки нужно сделать лишь пару кликов.) Реагенты тоже покупаются, но в перспективе будут создаваться и они.
В общем все это очень интересно, но главное – за этим будущее. Сегодня биотехнологии занимают в мировом научно-техническом прогрессе то же место, что компьютерные технологии в 80-х гг. прошлого века. При этом секвенирование – одно из ключевых направлений для современной биологии и медицины. Ну и, конечно, биотехнологии – это очень прибыльно.
В последнее время на рынке появился полупроводниковый секвенатор S5, и в ближайшее будущие я планирую переключиться на него.
Буду рад пообщаться со всеми, кто захочет тем или иным образом поучаствовать в развитии этого проекта!
Проект был бы не возможен, без теоретической подготовки Владимира Зубова. Выражаю ему свою благодарность.
Спасибо за внимание!